Гистоны

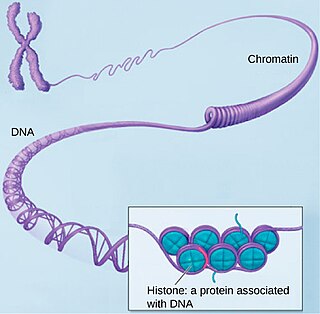
Гисто́ны (от греч. ἱστός «ткань») — обширный класс ядерных белков, выполняющих две основные функции: участие в упаковке нитей ДНК в ядре и эпигенетическая регуляция таких ядерных процессов, как транскрипция, репликация и репарация.
В хроматине гистоны составляют 25—40 % сухого веса[1]. Благодаря высокому содержанию лизина и аргинина гистоны проявляют сильно оснóвные свойства. Гистоны непосредственно контактируют с ДНК и способны нейтрализовать отрицательный заряд фосфатных групп ДНК за счёт положительных зарядов аминокислотных остатков. Последовательность аминокислот в этих белках является консервативной и практически не различается в организмах различных таксонов. Гистоны присутствуют в ядрах эукариотических клеток; у бактерий гистонов нет, но они выявлены у архей группы Euryarchaea[2].
Гистоны обнаружены в 1884 году немецким биохимиком Альбрехтом Косселем[3].
Типы гистонов и их роль
Существует всего пять различных типов гистонов H1/Н5, H2A, H2B, H3, H4.
Гистоны H2A, H2B, H3, H4, называемые кóровыми гистонами (от англ. core «сердцевина; ядро»), формируют нуклеосому, представляющую собой белковую глобулу, вокруг которой накручена нить ДНК. Центральная субъединица всех коровых гистонов имеет одинаковую вторичную структуру с протяжённым α-спиральным доменом, который с обеих сторон фланкируется доменами, содержащими по одной петле и по одной короткой α-спирали. Такая субъединица получила название «гистоновая складка»[4].
Все четыре типа коровых гистонов имеют одинаковую «гистоновую складку», при этом идентичность последовательностей между ними довольно низкая[5] (по некоторым оценкам, не превышает 25%).
Гистон H1/H5, называемый линкерным гистоном (от англ. link «связь»), связывается с внешней стороной нуклеосомы, фиксируя на ней нить ДНК. Участок ДНК, который следует за ним, называется линкерная ДНК (примерно 100 пар оснований). Гистон H1 является самым большим из всех гистонов. Он отличается от коровых гистонов и влияет на дальнейшую упаковку хроматина[4].
Нуклеосомы и линкерные гистоны имеют ряд функций, которые обуславливают динамику хроматина. Например, гистон H1 является фиксатором нити ДНК на нуклеосоме, и таким образом он контролирует доступность хроматина[6]. В свою очередь, коровые гистоны могут менять внутреннюю композицию и тем самым влиять на доступность хроматина к определенным участкам ДНК. Кроме того, нуклеосомные белки в ходе жизнедеятельности клетки подвергаются ряду посттрансляционных модификаций, включая ацетилирование, метилирование, фосфорилирование и убиквитилирование, которые могут изменять свойства и влиять на взаимодействие нуклеосомы с различными белками.
Гистоновые белки в структуре нуклеосомы

Нуклеосома преставляет из себя примерно 147 пар оснований (п.о.) ДНК, которая намотана вокруг октамера (~1,67 витка спирали), состоящего из пар белков, называемых коровыми гистонами. Диаметр ее составляет 7 нм. Длина фрагмента ДНК, "обхватывающего" одну нуклеосому, варьирует, в среднем составляет 200 п.о. При этом непосредственно с нуклеосомой связаны 146 п.о., а остальные несколько десятков соединяют две соседние нуклеосомы[7]. Линкерный гистон Н1 взаимодействует с линкерным участком ДНК, при этом не контактируя с гистоновым октамером.
Контакт между гистонами нуклеосомы и ДНК достаточно прочный. В каждой нуклеосоме между ДНК и гистонами, входящими в её состав, образуется 142 водородные связи. Почти половина этих связей возникает между основной цепью аминокислот гистонов и фосфодиэфирными группами сахарнофосфатного остова ДНК. Помимо водородных связей ДНК с белками, нуклеосомы скрепляют многочисленные гидрофобные взаимодействия и солевые мостики. Например, положительные заряды аминокислот лизина и аргинина, которыми обогащены гистоны, могут эффективно нейтрализовать отрицательный заряд остова ДНК. Эти многочисленные взаимодействия отчасти объясняют, почему ДНК практически любой последовательности может быть связана с нуклеосомным октамером[8].
Структура кóровых гистонов
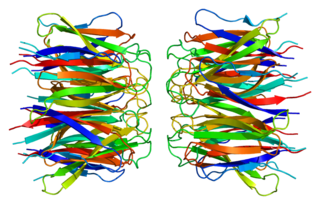
Кóровые гистоны Н2А, Н2В, Н3 и Н4 являются небольшими белками с молекулярными массами 10—15 кДа, состав которых чрезвычайно обогащён положительно заряженными аминокислотами лизином и аргинином[9]. Положительно заряженные аминокислоты сосредоточены в основном в аминных (N-) и карбоксильных (C-) (см. Пептидная связь) концевых частях молекул коровых гистонов, называемых хвостами. Гистоновые хвосты длиной около 15—30 аминокислотных остатков не организованы в какие-либо выраженные вторичные структуры. Гистоновые хвосты, прежде всего N-хвост, играют ключевую роль в эпигенетических механизмах, в которых участвуют эти белки. В центральных, самых консервативных, участках полипептидной цепи кóровых гистонов преобладают остатки гидрофобных аминокислот. Именно эти центральные области участвуют в образовании нуклеосомного октамера, на который навивается ДНК[3]. Центральная область всех нуклеосомных гистонов имеет характерную вторичную структуру с протяжённым α-спиральным доменом, который с обеих сторон фланкируется доменами, содержащими по одной петле и по одной короткой α-спирали. Эта пространственная структура называется гистоновой складкой (англ. histone fold domain, HFD)[10]. Таким образом, нуклеосомные гистоны содержат центральный структурированный трехспиральный HFD-домен и неструктурированные N- и C-хвосты.

Гистоны H3 и H4, Н2А и H2B попарно узнают друг друга. Спиральные домены взаимодействуют между собой, образуя структуры, названные рукопожатием, в результате чего возникают гетеродимеры — Н3-Н4 и Н2А-Н2В. Из первого димера, в свою очередь, образуется тетрамер (Н3-Н4)2. Тетрамер (Н3-Н4)2 и два димера Н2А-Н2В составляют гистоновый октамер, сердцевину нуклеосомы[3]. Нуклеосома имеет клиновидную форму. Её узкую часть составляет (Н3-Н4)2, а широкая часть состоит из двух димеров Н2А-Н2В, которые расположены по бокам тетрамера (Н3-Н4)2 и не взаимодействуют друг с другом. Из всей ДНК, что намотана на нуклеосомный октамер, примерно 80 пар оснований связаны с тетрамером (Н3-Н4)2 и около 40 пар с димерами Н2А-Н2В[10].
Структура линкерного гистона H1/Н5

С внешней стороной нуклеосомы в районе тетрамера (Н3-Н4)2 связывается линкерный гистон H1, фиксирующий тем самым на нуклеосоме нить ДНК. В эритроцитах птиц и рептилий в неактивном хроматине вместо гистона Н1 присутствует близкородственный гистон Н5[10]. Гистон Н1/Н5 существенно отличается от четырёх коровых гистонов. Он имеет молекулярную массу более 20 кДа. В его составе значительно больше остатков лизина, чем аргинина, причём все положительно заряженные аминокислотные остатки сконцентрированы на С-конце молекулы Н1. С-конец молекулы Н1 характеризуется неупорядоченной структурой и имеет длину около 100 аминокислотных остатков. Центральная часть молекулы Н1 богата гидрофобными аминокислотными остатками и в растворе образует глобулу. N-конец не имеет упорядоченной структуры и является относительно коротким[9].
Варианты гистонов и их роль
Одним из важных факторов регуляции работы эукариотической клетки на уровне нуклеосомы является замена гистонов на их варианты. Существует два вида гистонов: канонические и гистоновые варианты.
Все гистоны, кроме гистона H4, имеют соответствующие ему различные варианты. Канонические гистоны (H2A, H2B, H3, H4, H1/H5) обычно зависят от репликации[4]. Они особенно экспрессируются в S-фазе клеточного цикла. В то время как гистоновые варианты (H2A.Z, H2A.B, ..., H2B.W, H2B.Z, ..., H3.3, H3.Y, H3.5, ..., H1.0, H1.10) не зависят от репликации и экспрессируются в процессе всей жизнедеятельности клетки. Как канонические гистоны, так и их варианты, могут иметь свои отличительные особенности в зависимости от вида организма. Хотя, существуют и универсальные гистоны[11].
Роль гистоновых вариантов состоит в том, чтобы сохраняя нуклеосомную укладку хроматина, увеличивать или уменьшать её устойчивость, создавать особый контекст в каждом конкретном участке хроматина и тем самым управлять процессами транскрипции, репликации и репарации[10]. Каждый вариант гистона имеет характерную последовательность и структурные особенности, которые объясняют его специфическую функцию[4]. Причем, в то время как некоторые варианты могут отличаться всего парой аминокислотных остатков, другие же могут иметь более низкую схожесть. Например, H2B и H2B.E отличаются всего четырьмя или пятью аминокислотными остатками, а два подварианта H2A.Z (H2A.Z.1 и H2A.Z.2) у позвоночных - всего тремя. Аналогичная ситуация наблюдается и между гистоновым вариантом H2A.X и его канонической формой. Он отличается от H2A функционально важным C-терминальным мотивом фосфорилирования Ser-Gln- (Glu / Asp) -Ф, где Ф представляет собой гидрофобный остаток. Вариант-специфическое фосфорилирование серина в этом мотиве может происходить при образовании двухцепочечных разрывов ДНК и может иметь важное значение для вовлечения и удержания различных факторов ремоделирования хроматина, чтобы способствовать восстановлению двухцепочечных разрывов. В качестве примера пары вариантов с более низкой идентичностью можно привести H2A.L, идентичность последовательности которого с каноническим H2A составляет всего 24%[11].
Известно, что модификации над гистоновыми вариантами часто оказываются такими же, как и у их канонических форм. Например, Lys4 в H3.3 часто триметилирован (H3.3K4me3), а остатки Lys18 и Lys23 часто ацетилированы (H3.3K18ac и H3.3K23ac соответственно)[12].
Гены гистонов
Гены классических гистонов присутствуют в геноме в нескольких копиях и собраны в тандемно повторённые кластеры. Кластерная организация генов канонических гистонов характерна для всех многоклеточных. У человека самый большой кластер этих генов, называемый HIST1 и состоящий из 55 генов, находится на хромосоме 6 в участке 6p21-p22. Два кластера меньших размеров находятся на хромосоме 1: в бэнде 1q21 находится кластер HIST2, содержащий 6 гистоновых генов, а в бэнде 1q42 находится кластер HIST3, состоящий из трёх генов. Вне трёх вышеописанных кластеров на хромосоме 12 в бэнде 12p13.1 находится единственный ген, кодирующий канонический коровый гистон, — ген HIST4H4, кодирующий гистон H4[13].
Характерной чертой генов канонических гистонов является отсутствие интронов. Транскрипция этих генов происходит строго во время S-фазы клеточного цикла. Матричная РНК этих генов не полиаденилируется, 3'-некодирующая часть мРНК уложена во вторичную структуру типа «стебель-петля»[14].
В противоположность генам канонических гистонов гены вариантных гистонов не образуют кластеров, они разбросаны по всему геному, нередко содержат интроны, транскрибируемая с них РНК полиаденируется, транскрипция происходит во время всего клеточного цикла.
| Суперсемейство | Семейство | Подсемейство | Гены |
|---|---|---|---|
| Линкерный гистон | |||
| Гистон Н1 | |||
| Вариантные гистоны H1 (подсемейство H1F) | H1F0, H1FNT, H1FOO, H1FX, HILS1 | ||
| Гены канонического гистона H1 в кластере HIST1 (H1H1) | HIST1H1A, HIST1H1B, HIST1H1C, HIST1H1D, HIST1H1E, HIST1H1T | ||
| Коровые гистоны | |||
| Гистон H2A | |||
| Вариантные гистоны H2A (H2AF) | H2AFB1, H2AFB2, H2AFB3, H2AFJ, H2AFV, H2AFX, H2AFY, H2AFY2, H2AFZ | ||
| Гены канонического гистона H2A в кластере HIST1 (H2A1) | HIST1H2AA, HIST1H2AB, HIST1H2AC, HIST1H2AD, HIST1H2AE, HIST1H2AG, HIST1H2AI, HIST1H2AJ, HIST1H2AK, HIST1H2AL, HIST1H2AM | ||
| Гены канонического гистона H2A в кластере HIST2 (H2A2) | HIST2H2AA3, HIST2H2AC | ||
| Гистон H2B | |||
| Вариантные гистоны H2B (H2BF) | H2BFM, H2BFS, H2BFWT | ||
| Гены канонического гистона H2B в кластере HIST1 (H2B1) | HIST1H2BA, HIST1H2BB, HIST1H2BC, HIST1H2BD, HIST1H2BE, HIST1H2BF, HIST1H2BG, HIST1H2BH, HIST1H2BI, HIST1H2BJ, HIST1H2BK, HIST1H2BL, HIST1H2BM, HIST1H2BN, HIST1H2BO | ||
| Ген канонического гистона H2A в кластере HIST2 (H2B2) | HIST2H2BE | ||
| Гистон H3 | |||
| Гены канонического гистона H3 в кластере HIST1 (H3A1) | HIST1H3A, HIST1H3B, HIST1H3C, HIST1H3D, HIST1H3E, HIST1H3F, HIST1H3G, HIST1H3H, HIST1H3I, HIST1H3J | ||
| Гены канонического гистона H3 в кластере HIST2 (H3A2) | HIST2H3C | ||
| Гены канонического гистона H3 в кластере HIST3 (H3A3) | HIST3H3 | ||
| Гистон H4 | |||
| Гены канонического гистона H4 в кластере HIST1 (H41) | HIST1H4A, HIST1H4B, HIST1H4C, HIST1H4D, HIST1H4E, HIST1H4F, HIST1H4G, HIST1H4H, HIST1H4I, HIST1H4J, HIST1H4K, HIST1H4L | ||
| Ген канонического гистона H4 вне кластеров | HIST4H4 |
Модификации гистонов
Гистоны в октамере имеют подвижный N-концевой фрагмент («хвост») из 20 аминокислот, который выступает из нуклеосом и важен для поддержания структуры хроматина и контроля над генной экспрессией. Так, например, некоторые модификации гистонов (фосфорилирование и ацетилирование), как известно, локализованы преимущественно на участках хроматина с активными генами[15][16], тогда как их деацетилирование[17] и метилирование репрессорным комплексом поликомб играют важную роль в поддержании плюрипотентности и дифференцировке[18].
Детали механизма регуляции до конца не выяснены[19][20][21].
Консервативность гистонов
Последовательность аминокислот гистонов, то есть их первичная структура, мало изменилась в процессе эволюции. Это хорошо видно при сравнении аминокислотной последовательности гистонов млекопитающих, растений и дрожжей. Так, Н4 человека и пшеницы отличаются лишь несколькими аминокислотами. К тому же размер молекулы белка и её полярность довольно постоянны. Из этого можно заключить, что гистоны были оптимизированы ещё в эпоху общего предшественника животных, растений и грибов (более 700 млн лет назад). Хотя с тех пор в гистоновых генах происходили бесчисленные точечные мутации, все они, очевидно, приводили к вымиранию мутантных организмов.
См. также
- Белки группы polycomb
- CAF-1
- Хроматин
- Подавление экспрессии генов
- Гистонацетилтрансфераза
- Деацетилазы гистонов
- Нуклеосома
Примечания
- ↑ Биологический энциклопедический словарь / Гл.ред. М.С.Гиляров. — М.: Сов. энциклопедия, 1986. — 831 с.
- ↑ Нуклеиновые кислоты: от А до Я / Б. Аппель [и др.]. — М.: Бином: Лаборатория знаний, 2013. — 413 с. — 700 экз. — ISBN 978-5-9963-0376-2.
- ↑ 1 2 3 Карпов В.Л. От чего зависит судьба гена // Природа. — Наука, 2005. — № 3. — С. 34—43. Архивировано 10 февраля 2018 года.
- ↑ 1 2 3 4 Eli J. Draizen, Alexey K. Shaytan, Leonardo Mariño-Ramírez, Paul B. Talbert, David Landsman. HistoneDB 2.0: a histone database with variants—an integrated resource to explore histones and their variants (англ.) // Database. — 2016. — Vol. 2016. — P. baw014. — ISSN 1758-0463. — doi:10.1093/database/baw014. Архивировано 19 января 2022 года.
- ↑ Andreas D. Baxevanis, Gina Arents, Evangelos N. Moudrianakis, David Landsman. A variety of DNA-binding and multimeric proteins contain the histone fold motif (англ.) // Nucleic Acids Research. — 1995. — Vol. 23, iss. 14. — P. 2685–2691. — ISSN 1362-4962 0305-1048, 1362-4962. — doi:10.1093/nar/23.14.2685.
- ↑ Grigoriy A Armeev, Anna K Gribkova, Iunona Pospelova, Galina A Komarova, Alexey K Shaytan. Linking chromatin composition and structural dynamics at the nucleosome level (англ.) // Current Opinion in Structural Biology. — 2019-06. — Vol. 56. — P. 46–55. — doi:10.1016/j.sbi.2018.11.006. Архивировано 14 июня 2022 года.
- ↑ Коряков Д. Е. Модификации гистонов и регуляция работы хроматина // Генетика. — 2006. — Т. 42, № 9. — С. 1170—1185.
- ↑ Молекулярная биология клетки: в 3-х томах / Б. Альбертс, А. Джонсон, Д. Льюис и др. — М.-Ижевск: НИЦ «Регулярная и хаотическая динамика», Институт компьютерных исследований, 2013. — Т. I. — С. 325—359. — 808 с. — ISBN 978-5-4344-0112-8.
- ↑ 1 2 Разин С. В. Хроматин: упакованный геном / С. В. Разин, А. А. Быстрицкий. — М.: БИНОМ: Лаборатория знаний, 2009. — С. 4-8. — 176 с. — ISBN 978-5-9963-0087-7.
- ↑ 1 2 3 4 Коряков Д. Е. Нуклеосомная организация хроматина // Эпигенетика / С. М. Закиян, В.В. Власов, Е. В. Дементьева. — Новосибирск: Изд-во СО РАН, 2012. — С. 7—30. — 592 с. — 300 экз. — ISBN 978-5-7692-1227-7.
- ↑ 1 2 Alexey K Shaytan, David Landsman, Anna R Panchenko. Nucleosome adaptability conferred by sequence and structural variations in histone H2A–H2B dimers (англ.) // Current Opinion in Structural Biology. — 2015-06. — Vol. 32. — P. 48–57. — doi:10.1016/j.sbi.2015.02.004. Архивировано 8 марта 2022 года.
- ↑ Paul B. Talbert, Steven Henikoff. Histone variants on the move: substrates for chromatin dynamics (англ.) // Nature Reviews Molecular Cell Biology. — 2017-02. — Vol. 18, iss. 2. — P. 115–126. — ISSN 1471-0080 1471-0072, 1471-0080. — doi:10.1038/nrm.2016.148. Архивировано 3 июня 2022 года.
- ↑ Marzluff W. F., Gongidi P., Woods K. R., Jin J., Maltais L. J. The human and mouse replication-dependent histone genes (англ.) // Genomics : journal. — Academic Press, 2002. — November (vol. 80, no. 5). — P. 487—498. — PMID 12408966. Архивировано 5 марта 2016 года. Архивированная копия. Дата обращения: 14 июля 2013. Архивировано 5 марта 2016 года.
- ↑ Marzluff W. F., Wagner E. J., Duronio R. J. Metabolism and regulation of canonical histone mRNAs: life without a poly(A) tail (англ.) // Nat. Rev. Genet. : journal. — 2008. — November (vol. 9, no. 11). — P. 843—854. — doi:10.1038/nrg2438. — PMID 18927579. — PMC 2715827. Архивировано 30 мая 2016 года.
- ↑ Zheng Y. et al. Histone H1 phosphorylation is associated with transcription by RNA polymerases I and II (англ.) // The Journal of Cell Biology. — 2010. — Vol. 189, iss. 3. — P. 407. — doi:10.1083/jcb.201001148.
- ↑ Creyghton M. P. et al. Histone H3K27ac separates active from poised enhancers and predicts developmental state (англ.) // Proc Natl Acad Sci U S A. — 2010. — Vol. 107, iss. 50. — P. 21931—21936. — doi:10.1073/pnas.1016071107.
- ↑ Guang Hu, Paul A. Wade. NuRD and Pluripotency: A Complex Balancing Act (англ.) // Cell Stem Cell. — 2012. — Vol. 10, iss. 5. — P. 497—503. — doi:10.1016/j.stem.2012.04.011. Архивировано 24 сентября 2015 года.
- ↑ Gerasimova A. et al. Predicting Cell Types and Genetic Variations Contributing to Disease by Combining GWAS and Epigenetic Data (англ.) // PLOS One. — Public Library of Science, 2013. — Vol. 8, iss. 1. — P. e54359. — doi:10.1371/journal.pone.0054359.
- ↑ Pengelly A. R. et al. A Histone Mutant Reproduces the Phenotype Caused by Loss of Histone-Modifying Factor Polycomb (англ.) // Science. — 2013. — Vol. 339, iss. 6120. — P. 698. — doi:10.1126/science.1231382.
- ↑ Histone Modification Controls Development: Chemical Tags On Histones Regulate Gene Activity. Дата обращения: 12 февраля 2013. Архивировано 11 февраля 2013 года.
- ↑ Moyra Lawrence, Sylvain Daujat, Robert Schneider. How Histone Modifications Regulate Gene Expression (англ.) // Trends in Genetics. — Cell Press, 2015. — Vol. 32, iss. 1. — P. 42—56. — doi:10.1016/j.tig.2015.10.007.
Ссылки
- HistoneDB 2.0 — Database of histones and variants at NCBI (англ.)
- Chromatin, Histones & Cathepsin; the proteolysis map (PMAP) — animation (англ.)