Пирролизин
| Пирролизин | |
|---|---|
  | |
| Общие | |
| Хим. формула | C12H21N3O3 |
| Физические свойства | |
| Молярная масса | 255,313 г/моль |
| Классификация | |
| Рег. номер CAS | 448235-52-7 |
| PubChem | 5460671 и 23657811 |
| SMILES | |
| InChI | |
| ChEBI | 21860 и 58499 |
| ChemSpider | 4574156 |
| Приведены данные для стандартных условий (25 °C, 100 кПа), если не указано иное. | |
Пирролизин — нестандартная протеиногенная аминокислота, которая участвует в биосинтезе белка у ряда метаногенных архей [1]. Был обнаружен в 2002 году на активном участке фермента метилтрансферазы, выделенной из метаногенной археи Methanosarcina barkeri[2][3], и является 22-й из найденных в природе аминокислот, которые входят в состав природных белков [4][5]. В организме человека отсутствует.
Для обозначения пирролизина ИЮПАК рекомендует трёхбуквенное сокращение Pyl и однобуквенное O [6].
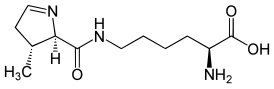
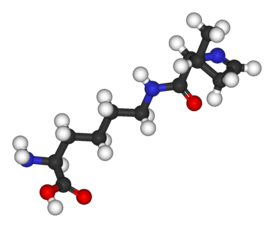
Пирролизин содержит в своей структуре карбоксильную группу и аминогруппу в α-положении к ней; в живых организмах существует в цвиттер-ионной форме. Имеет структурную аналогию с лизином по наличию азотсодержащего осно́вного фрагмента в 6-м положении углеводородной цепи.
Строение
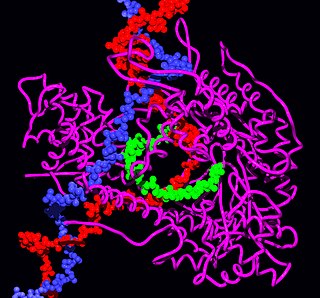
Согласно данным рентгеноструктурного анализа[3] и MALDI масс-спектрометрии, пирролизин является производным лизина, содержащим (4r, 5r)-4-пирролин-5-карбоксилатный заместитель, соединенный с ϵ-атомом азота пептидной связью[7].
Синтез
Синтез пирролизина in vivo протекает путем соединения двух молекул L-лизина с участием фермента пирролизил-тРНК-синтазы[8]. Одна молекула лизина сначала изомеризуется в (R)-3-метил-D-орнитин, который далее образует пептидную связь со второй молекулой лизина, с последующей циклизацией с удалением аминогруппы и дегидрированием с образованием пирролинового фрагмента[9].
Химический синтез пирролизина включает ацилирование по ε-аминогруппе лизина, предварительно защищенного по α-NH2-группе, под действием (4R, 5R)-4-метилпирролин-5-карбоновой кислоты [10].
Генетическое кодирование
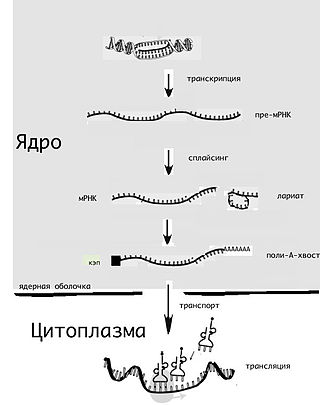
В биосинтезе белков у архей включение пирролизина в полипептидную цепь закодировано в мРНК кодоном UAG (урацил–аденин–гуанин), который для клеток большинства организмов является стоп-кодоном, т.е. не предполагает включение рибосомой в синтезируемую ей петидную цепь белка какой-либо аминокислоты – связывающаяся со стоп-кодоном тРНК поступает в рибосому без аминокислоты, что, как правило, влечет за собой обрыв полипетидной цепи и завершение синтеза белковой молекулы. Особенность трансляции кодона UAG в клетках архей обусловлено наличием у них гена pylT, который кодирует синтез необычной тРНК с антикодоном CUA – т-РНКCUA (кодон CUA, цитозин–урацил–аденин, для большинства организмов кодирует лизин), и гена pylS, который кодирует энзим pylRS, разновидность характерной для архей аминоацил-тРНК-синтазы – класса ферментов, ответственных за присоединение аминокислот к тРНК. pylRS катализирует присоединение именно пирролизина, а не лизина к нетипичной тРНКCUA архей, которая доставляет его в рибосому за счет своей необычной способности присоединяться к участку мРНК с кодоном UAG и включает в процесс биосинтеза белка[2][11].
Оперон, содержащий гены pylT и pylS, найден при секвенировании геномов всех известных представителей архей из семейства Methanosarcinaceae. Гомологи генов pylS и pylT обнаружены также в геноме грам-положительной бактерии Desulfitobacterium hafniense, хотя функции этих гомологов у данной бактерии неизвестны[12][13]. Введение генов pylS и pylT в клетку e-coli привели к переопределению у неё функции UAG со стоп-кодона на кодон, отвечающий за включение в синтезируемый белок молекулы пирролизина, после чего бактерия получила возможность к использованию экзогенного пирролизина в составе синтезируемых ею белков. Это подтвердило, что использование пирролизина в биосинтезе белка в ответ на UAG кодон имеет генетические причины, т.е. определяется наличием у организма соответствующих генов [11][14].
Наличие генов pylT и pylS определяет использования пирролизина как результат трансляции кодона UAG с высокой селективностью. Так, при этом невозможно использовать вместо пирролизина его структурный аналог лизин[15], а многие ε-N-ацилпроизводные лизина удалось вовлечь в биосинтез белка в незначительных количествах[15][16]. Однако разработаны аналоги лизина, например, с заменой пирролинового кольца на тетрагидрофурановое[15][17], способные при наличии генов pylT и pylS к связыванию с тРНКCUA и включению в значительных колиечествах в синтезируемый белок. Считается, что эффективные аналоги пирролизина при этом должны иметь в своей структуре вместо пирролинового кольца пятичленный гетероцилический заместитель с гетероатомом во 2-м положении[17].
Каталитическая функция
Фрагмент пирролизина входит в состав активного центра нескольких метилтрансфераз архей – ферментов, участвующих в процессе метаногенеза, обеспечивающего клетки архей энергией, и ответственных за процесс связывания N-метиламинов и отщепление их метильной группы, которая превращается в метан в процессе дальнейшего метаболизма. Считается, что конформационная подвижность пирролинового фрагмента пирролизина в структуре этих ферментов достаточно высока, и именно его иминная связь играет решающую роль в процессе расщепления связи С–N метиламина, захватываемого и удерживаемого внутри активного центра фрагментами двух других аминокислот – тирозина и глутаминовой кислоты. При этом метиламин присоединяется по двойной связи пирролинового цикла с образованием 1,1-диаминопроизводного, связь С–N метиламинового остатка гетеролитически расщепляется корриноидным кофактором, образующийся метилкатион связывается с атомом кобальта кофактора (с изменением степени окисления Co с +1 до +3) и передаётся далее в метаболическую цепь. Оставшаяся связанной с ферментом аминогруппа захватывает Н+ из внешней среды и выводится в составе аммиака, а пирролиновый фрагмент фермента далее участвует в следующем каталитическом цикле[3].
См. также
- Генетический код
- Трансляция
- Селеноцистеин, 21-я аминокислота. Как и пирролизин, закодирована стоп-кодоном.
Примечания
- ↑ Michael Rother, Joseph A. Krzycki. Selenocysteine, Pyrrolysine, and the Unique Energy Metabolism of Methanogenic Archaea (англ.) // Archaea. — 2010. — Vol. 2010. — P. 1–14. — ISSN 1472-3654 1472-3646, 1472-3654. — doi:10.1155/2010/453642. Архивировано 7 февраля 2021 года.
- ↑ 1 2 Gayathri Srinivasan, Carey M. James, Joseph A. Krzycki. Pyrrolysine Encoded by UAG in Archaea: Charging of a UAG-Decoding Specialized tRNA (англ.) // Science. — 2002-05-24. — Vol. 296, iss. 5572. — P. 1459–1462. — ISSN 1095-9203 0036-8075, 1095-9203. — doi:10.1126/science.1069588. Архивировано 18 мая 2023 года.
- ↑ 1 2 3 B. Hao. A New UAG-Encoded Residue in the Structure of a Methanogen Methyltransferase (англ.) // Science. — 2002-05-24. — Vol. 296, iss. 5572. — P. 1462–1466. — doi:10.1126/science.1069556.
- ↑ Amanda Yarnell. 22ND AMINO ACID IDENTIFIED: Methanogen uses stop codon to genetically encode L-pyrrolysine (англ.) // Chemical & Engineering News Archive. — 2002-05-27. — Vol. 80, iss. 21. — P. 13. — ISSN 2157-4936 0009-2347, 2157-4936. — doi:10.1021/cen-v080n021.p013. Архивировано 18 мая 2023 года.
- ↑ John F. Atkins, Ray Gesteland. The 22nd Amino Acid (англ.) // Science. — 2002-05-24. — Vol. 296, iss. 5572. — P. 1409–1410. — ISSN 1095-9203 0036-8075, 1095-9203. — doi:10.1126/science.1073339. Архивировано 18 мая 2023 года.
- ↑ Nomenclature and symbolism for amino acids and peptides (Recommendations 1983) (англ.) // Pure and Applied Chemistry. — 1984-01-01. — Vol. 56, iss. 5. — P. 595–624. — ISSN 0033-4545 1365-3075, 0033-4545. — doi:10.1351/pac198456050595. Архивировано 18 мая 2023 года.
- ↑ Jitesh A. Soares, Liwen Zhang, Rhonda L. Pitsch, Nanette M. Kleinholz, R. Benjamin Jones. The Residue Mass of L-Pyrrolysine in Three Distinct Methylamine Methyltransferases // Journal of Biological Chemistry. — 2005-11. — Т. 280, вып. 44. — С. 36962–36969. — ISSN 0021-9258. — doi:10.1074/jbc.m506402200.
- ↑ Tatsuo Yanagisawa, Tomomi Sumida, Ryohei Ishii, Shigeyuki Yokoyama. A novel crystal form of pyrrolysyl-tRNA synthetase reveals the pre- and post-aminoacyl-tRNA synthesis conformational states of the adenylate and aminoacyl moieties and an asparagine residue in the catalytic site (англ.) // Acta Crystallographica Section D - Structural Biology : журнал. — 2013. — 15 January (no. 69). — P. 5 - 15. — ISSN 2059-7983. Архивировано 23 февраля 2023 года.
- ↑ Marsha A. Gaston, Liwen Zhang, Kari B. Green-Church, Joseph A. Krzycki. The complete biosynthesis of the genetically encoded amino acid pyrrolysine from lysine (англ.) // Nature. — 2011-03. — Vol. 471, iss. 7340. — P. 647–650. — ISSN 1476-4687 0028-0836, 1476-4687. — doi:10.1038/nature09918. Архивировано 28 января 2022 года.
- ↑ Bing Hao, Gang Zhao, Patrick T. Kang, Jitesh A. Soares, Tsuneo K. Ferguson, Judith Gallucci, Joseph A. Krzycki, Michael K. Chan. Reactivity and Chemical Synthesis of L-Pyrrolysine— the 22nd Genetically Encoded Amino Acid (англ.) // Chemistry & Biology. — 2004-09. — Vol. 11, iss. 9. — P. 1317–1324. — doi:10.1016/j.chembiol.2004.07.011. Архивировано 13 августа 2022 года.
- ↑ 1 2 Sherry K. Blight, Ross C. Larue, Anirban Mahapatra, David G. Longstaff, Edward Chang, Gang Zhao, Patrick T. Kang, Kari B. Green-Church, Michael K. Chan, Joseph A. Krzycki. Direct charging of tRNACUA with pyrrolysine in vitro and in vivo (англ.) // Nature. — 2004-09. — Vol. 431, iss. 7006. — P. 333–335. — ISSN 1476-4687. — doi:10.1038/nature02895. Архивировано 14 февраля 2023 года.
- ↑ John F. Atkins and Ray Gesteland. The 22nd Amino Acid (англ.) // Science. — 2002. — Vol. 296, no. 5572. — P. 1409—1410. — doi:10.1126/science.1073339. — PMID 12029118.
- ↑ S. Herring, A. Ambrogelly, C. R. Polycarpo, D. Soll. Recognition of pyrrolysine tRNA by the Desulfitobacterium hafniense pyrrolysyl-tRNA synthetase (англ.) // Nucleic Acids Research. — 2007-01-30. — Vol. 35, iss. 4. — P. 1270–1278. — ISSN 1362-4962 0305-1048, 1362-4962. — doi:10.1093/nar/gkl1151. Архивировано 5 апреля 2023 года.
- ↑ J. Krzycki. The direct genetic encoding of pyrrolysine (англ.) // Curr Opin Microbiol. — 2005. — Vol. 8, no. 6. — P. 706—712. — doi:10.1016/j.mib.2005.10.009. — PMID 16256420.
- ↑ 1 2 3 Wen-Tai Li, Anirban Mahapatra, David G. Longstaff, Jonathan Bechtel, Gang Zhao, Patrick T. Kang, Michael K. Chan, Joseph A. Krzycki. Specificity of Pyrrolysyl-tRNA Synthetase for Pyrrolysine and Pyrrolysine Analogs (англ.) // Journal of Molecular Biology. — 2009-01. — Vol. 385, iss. 4. — P. 1156–1164. — doi:10.1016/j.jmb.2008.11.032. Архивировано 13 августа 2022 года.
- ↑ Takahito Mukai, Takatsugu Kobayashi, Nobumasa Hino, Tatsuo Yanagisawa, Kensaku Sakamoto, Shigeyuki Yokoyama. Adding l-lysine derivatives to the genetic code of mammalian cells with engineered pyrrolysyl-tRNA synthetases (англ.) // Biochemical and Biophysical Research Communications. — 2008-07. — Vol. 371, iss. 4. — P. 818–822. — doi:10.1016/j.bbrc.2008.04.164. Архивировано 13 августа 2022 года.
- ↑ 1 2 Tomasz Fekner, Xin Li, Marianne M. Lee, Michael K. Chan. A Pyrrolysine Analogue for Protein Click Chemistry (англ.) // Angewandte Chemie International Edition. — 2009-01-20. — Vol. 48, iss. 9. — P. 1633–1635. — doi:10.1002/anie.200805420. Архивировано 19 мая 2023 года.