Pandoravirus
| Pandoravirus | |
|---|---|
 | |
| Научная классификация | |
| Международное научное название | |
| Pandoravirus | |
| Виды[3] | |
| |
| Группа по Балтимору | |
| I: дцДНК-вирусы | |
Pandoravirus (лат.) — род вирусов из монотипного семейства Pandoraviridae. Включает 6 видов. Один из крупнейших известных вирусов (на момент открытия — самый крупный).
По состоянию на октябрь 2018 года семейство, род и виды Pandoravirus не зарегистрированы в базе данных Международного комитета по таксономии вирусов (ICTV)[4].
История изучения
Пандоравирусы были открыты в 2013 году[5] при систематическом исследовании образцов воды и донных отложений в поисках новых вирусов семейства Mimiviridae[англ.], поражающих амёб Acantamoeba[англ.]. Как и мимивирусы, они были выделены из образцов, которые демонстрировали сильную литическую активность при совместном выращивании с культурами[англ.] Acanthamoeba. Первый вид нового рода, Pandoravirus salinus, был выделен из образца неглубоких донных отложений из побережья центрального Чили, а второй вид, Pandoravirus dulcis, был изолирован из грязи, собранной со дна пресного пруда рядом с Мельбурном (Австралия). Геномы обоих вирусов были секвенированы при их выделении. Ещё один вирус, имеющий морфологию Pandoravirus, Pandoravirus inopinatum, был выделен в 2008 году у пациента, страдавшего от кератита, вызванного Acanthamoeba. Только спустя шесть лет из-за большого размера удалось установить его вирусную природу. В 2015 году его геном был секвенирован[6]. В 2018 году описано ещё три вида: Pandoravirus macleodensis найден в образцах пресного водоёма рядом с Мельбурном, всего в 700 м от того места, где найден P. dulcis; Pandoravirus neocaledonia изолирован в солоноватой воде мангровых зарослей возле аэропорта Нумеа (Новая Каледония) и Pandoravirus quercus, найденный в грунте Марселя (Франция). Тогда же принято решение выделить род в отдельное семейство. Результат филогенетического исследования рода, проведённого при описании этих трёх видов, можно проиллюстрировать следующей кладограммой[3]:
| Pandoravirus |
| ||||||||||||||||||||||||||||||
Структура
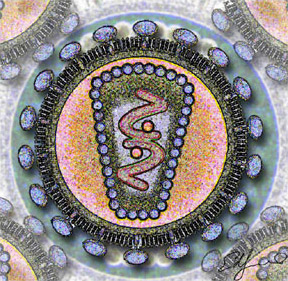
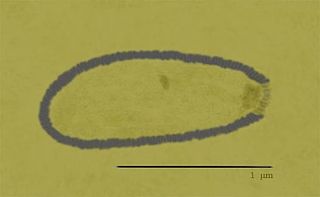
Pandoravirus можно рассмотреть в световой микроскоп. Вирионы представляют собой яйцевидные частицы 0,8—1,2 мкм длиной и 0,5 мкм в диаметре. Электронная микроскопия выявила уникальную структуру этих вирионов, не встречающуюся больше ни у каких описанных вирусов. Зрелый вирион представляет собой выглядящий пустым компартмент, окружённый мембраной, под которой располагается оболочка толщиной около 70 нм. В ней выделяются три слоя: внутренний электронно-плотный слой толщиной около 20 нм, средний тёмный слой толщиной около 25 нм, похожий на густую сеть из параллельно ориентированных фибрилл, и наружный слой средней электронной плотности толщиной около 25 нм. На одном конце вирусной частицы находится апикальная пора, через которую неохарактеризованное содержимое вириона изливается в цитоплазму амёбы-хозяина, проходя через канал, образованный при слиянии мембраны вируса и мембраны пищеварительной вакуоли. В отличие от мимивируса, у пандоравирусов нет электронно-плотной центральной области, которая соответствует компактизированному геному[6].
Геном
Геном пандоравирусов представлен линейной двуцепочечной ДНК длиной 2,77 млн пар оснований у P. salinus, 1,93 млн пар оснований у P. dulcis и 2,24 млн пар оснований у P. inopinatum[6]. GC-состав пандоравирусных геномов оказался высоким: 61,7, 63,7 и 60,7 % у P. salinus, P. dulcis и P. inopinatum соответственно (наибольший известный GC-состав у вируса характерен для герпесвирусов: более 70 %). У P. inopinatum обнаружено 89 % общих последовательностей с P. dulcis и 85 % с P. salinus. Размер капсида и молекулы ДНК, а также её плотность укладки говорят о том, что геномная ДНК P. salinus без труда помещается в их капсиде. В геноме P. salinus обнаружено 2556 предполагаемых белок-кодирующих генов, у P. dulcis — 1502, у P. inopinatum — 1339. Состав генов у P. salinus и P. dulcis примерно одинаков, различия в размере генома обусловлены четырьмя сегментами, уникальными для P. salinus. Показано, что в геноме P. salinus имеется множество транспозонов[7]. Предполагаемые белки, кодируемые геномом P. salinus, имеют размер от 26 до 2367 аминокислотных остатков (а. о.), средняя длина составляет 258 а. о.[5][8] У трёх видов, описанных в 2018 году — P. neocaledonia, P. macleodensis и P. quercus — геном также представлен линейной двуцепочечной молекулой ДНК, включающей от 1,84 до 2 млн пар оснований. Кроме того, как и у ранее известных видов рода Pandoravirus, их геномы также имеют очень высокий GC-состав — около 60 %[3].
Жизненный цикл
Жизненный цикл пандоравирусов в амёбе Acanthamoeba castellanii длится от 10 до 15 часов и начинается с попадания вириона внутрь пищеварительной вакуоли, сформировавшейся при фагоцитозе вирусной частицы. Далее пандоравирус изливает внутрь клетки своё содержимое через апикальную пору. Через канал, образовавшийся при слиянии мембран вируса и пищеварительной вакуоли, в цитоплазму амёбы попадает вирусная ДНК и белки. После этого содержимое вируса в цитоплазме становится невидимым. Через 4 часа ядро амёбы начинает сильно реорганизовываться и теряет свою сферическую форму. Электронно-плотное ядрышко постепенно исчезает, а на ядерной мембране формируется множество впячиваний, образующих многочисленные везикулы. На периферии растворяющегося ядра появляется похожая на пероксисому кристаллическая структура, исчезающая по мере созревания вирусной частицы. Спустя 8—10 часов после начала инфекции клетки округляются и открепляются от субстрата, и на периферии, там, где когда-то было ядро, появляются вирусные частицы. В отличие от других ДНК-содержащих вирусов эукариот и фагов, у которых сначала образуется капсид, а потом он наполняется необходимым содержимым, у пандоравирусов оба процесса идут одновременно. Любопытно, что образование капсида начинается с той верхушки, которая несёт апикальную пору. Репликационный цикл завершается, когда клетка амёбы лизируется и высвобождает наружу сотню вирусных частиц[5].
Примечания
- ↑ Таксон не признан Международным комитетом по таксономии вирусов (ICTV).
- ↑ Таксономия вирусов (англ.) на сайте Международного комитета по таксономии вирусов (ICTV).
- ↑ 1 2 3 Legendre M., Fabre E., Poirot O., Jeudy S., Lartigue A., Alempic J. M., Beucher L., Philippe N., Bertaux L., Christo-Foroux E., Labadie K., Couté Y., Abergel C., Claverie J. M. Diversity and evolution of the emerging Pandoraviridae family (англ.) // Nature Communications. — 2018. — 11 June (vol. 9, no. 1). — ISSN 2041-1723. — doi:10.1038/s41467-018-04698-4.
- ↑ Search Pandoravirus in ICTV database. Дата обращения: 11 ноября 2018. Архивировано из оригинала 4 октября 2013 года.
- ↑ 1 2 3 Philippe N., Legendre M., Doutre G., Couté Y., Poirot O., Lescot M., Arslan D., Seltzer V., Bertaux L., Bruley C., Garin J., Claverie J. M., Abergel C. Pandoraviruses: amoeba viruses with genomes up to 2.5 Mb reaching that of parasitic eukaryotes. (англ.) // Science (New York, N.Y.). — 2013. — Vol. 341, no. 6143. — P. 281—286. — doi:10.1126/science.1239181. — PMID 23869018.
- ↑ 1 2 3 Abergel C., Legendre M., Claverie J. M. The Rapidly Expanding Universe of Giant Viruses: Mimivirus, Pandoravirus, Pithovirus and Mollivirus // FEMS Microbiology Reviews. — 2015. — Vol. 39, no. 6. — P. 779—796. — doi:10.1093/femsre/fuv037. — PMID 26391910.
- ↑ Sun C., Feschotte C., Wu Z., Mueller R. L. DNA transposons have colonized the genome of the giant virus Pandoravirus salinus. (англ.) // BMC biology. — 2015. — Vol. 13. — P. 38. — doi:10.1186/s12915-015-0145-1. — PMID 26067596.
- ↑ Antwerpen M. H., Georgi E., Zoeller L., Woelfel R., Stoecker K., Scheid P. Whole-genome sequencing of a pandoravirus isolated from keratitis-inducing acanthamoeba. (англ.) // Genome announcements. — 2015. — Vol. 3, no. 2. — doi:10.1128/genomeA.00136-15. — PMID 25814595.