Хими́ческая фо́рмула — условное обозначение химического состава и структуры соединений с помощью символов химических элементов, числовых и вспомогательных знаков. Химические формулы являются составной частью языка химии, на их основе составляются схемы и уравнения химических реакций, а также химическая классификация и номенклатура веществ. Одним из первых начал использовать их русский химик А. А. Иовский.
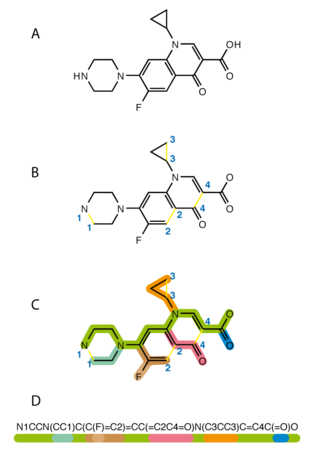
SMILES — система правил (спецификация) однозначного описания состава и структуры молекулы химического вещества с использованием строки символов ASCII. Название в английском языке является омонимом к слову smiles (улы́бки), однако пишется только заглавными буквами. В русском языке однозначного аналога не имеет, рекомендуется употребление на языке оригинала. Произносится как «смайлз».
Хемоинформатика — применение методов информатики для решения химических проблем.
Молекулярное моделирование (ММ) — собирательное название методов исследования структуры и свойств молекул вычислительными методами с последующей визуализацией результатов, обеспечивающие их трехмерное представления при заданных в расчете условиях.
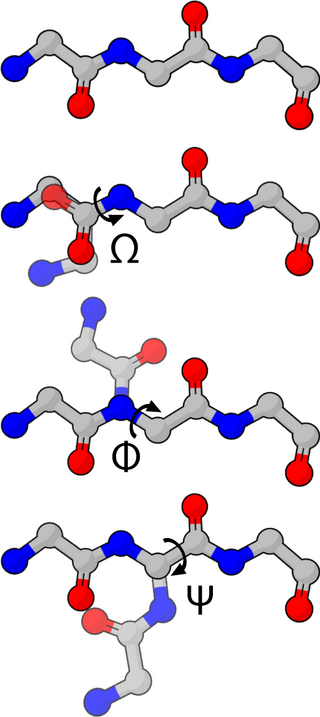
В химии Z-матрицей (Z-matrix) называют способ представления координат атомов молекулярной системы. Кроме того, такое представление называют также внутренними координатами. Это представление определяет каждый атом системы через атомный номер, длину связи, валентный угол и двугранный угол. Под связью в данном случае подразумевается не химическая связь, а просто вектор, направленный от одного атома к другому, хотя они могут и совпадать. Тем не менее, принято записывать Z-матрицу через длины и углы химических связей, так как такая запись позволяет описать не только относительное расположение атомов друг относительно друга, но и связи этих атомов. Z-матрица называется так потому, что второй атом всегда располагается вдоль оси аппликат.

Бактериородопси́ны — семейство мембранных светочувствительных белков археот. Бактериородопсины осуществляют перенос протона через плазматическую мембрану, по строению сходны с родопсинами млекопитающих.

Молекуля́рный до́кинг — метод молекулярного моделирования, позволяющий предсказать наиболее выгодную для образования устойчивого комплекса ориентацию и конформацию одной молекулы (лиганда) в сайте связывания другой (рецептора). Данные о положении и конформации партнеров используются для предсказания силы взаимодействия посредством так называемых оценочных функций. В случае, если лиганд является макромолекулой, докинг называют макромолекулярным.
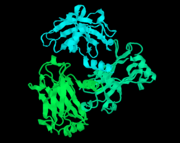
Protein Data Bank, PDB — банк данных трёхмерных структур белков и нуклеиновых кислот. Информация, полученная методами рентгеновской кристаллографии или ЯМР-спектроскопии, и, всё чаще, методом криоэлектронной микроскопии вносится в базу данных биологами и биохимиками со всего мира, и доступна бесплатно через интернет сайты организаций-членов.

Jmol — программа для просмотра структуры молекул в трёх измерениях.

OpenBabel — свободная химическая экспертная система, в основном используемая для преобразования форматов файлов. Эта программа доступна для UNIX и Windows и распространяется на условиях GNU GPL.
Выра́внивание после́довательностей — биоинформатический метод, основанный на размещении двух или более последовательностей мономеров ДНК, РНК или белков друг под другом таким образом, чтобы легко увидеть сходные участки в этих последовательностях. Сходство первичных структур двух молекул может отражать их функциональные, структурные или эволюционные взаимосвязи. Выровненные последовательности оснований нуклеотидов или аминокислот обычно представляются в виде строк матрицы. Добавляются разрывы между основаниями таким образом, чтобы одинаковые или похожие элементы были расположены в следующих друг за другом столбцах матрицы.
В химии, раскраска по CPK — популярная схема цветового обозначения атомов различных химических элементов в молекулярных моделях. Данная схема названа в честь молекулярной модели CPK, созданной Робертом Кори и Лайнусом Полингом и усовершенствованной Уолтером Колтуном.

Простра́нственное выра́внивание — способ установления гомологии между двумя или более полимерными структурами на основании их трёхмерной структуры. Этот процесс обычно применяется к третичной структуре белков, но может также использоваться и для больших молекул РНК. В противоположность простому наложению структур, когда известно по крайней мере несколько эквивалентных аминокислотных остатков, пространственное выравнивание не требует никаких предварительных данных, кроме координат атомов.

PyMOL — свободная система визуализации молекул. Позволяет создавать высококачественные трёхмерные изображения мелких молекул и биологических макромолекул, таких как белки. Примерно четверть всех публикуемых структур белков в научной литературе сделана с помощью PyMOL.

AutoDock — пакет программ, предназначенный для автоматизированного молекулярного докинга. В основном применяется для белок-лигандного докинга, в том числе, с учётом подвижных остатков белка. Autodock также используется для «слепого докинга», когда активный центр белка не известен.
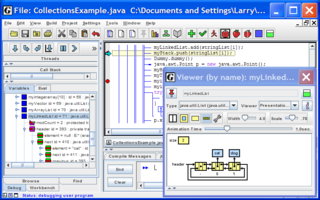
jGRASP — лёгкая интегрированная среда разработки, созданная специально для обеспечения автоматического создания визуализации работы приложения с целью улучшения его понятливости. Она создаёт статическую визуализацию структуры исходного кода программы и визуализацию структур данных во время исполнения скомпилированного кода.

Научная визуализация — это междисциплинарная отрасль науки. Согласно Фриндли, она «главным образом имеет дело с визуализацией трёхмерных явлений, при этом акцент делается на реалистичное изображение объёмов, поверхностей, источников освещения и так далее, возможно, в динамике ». Научная визуализация рассматривает также подмножество методов компьютерной графики, раздела информатики. Целью научной визуализации является графическая иллюстрация научных данных для возможности научным работникам понять, просмотреть и получить представление о данных.

Аконитаза, также аконитатгидратаза — фермент, из семейства гидратазы, который катализирует стереоспецифическую реакцию изомеризации цитрата в изоцитрат, посредством образования цис-аконитата в цикле Кребса, без окислительно-восстановительного механизма.
В химии простейшая формула химического соединения — формула с простейшим положительным целочисленным соотношением атомов, присутствующих в соединении. Простым примером этой концепции является то, что простейшая формула монооксида серы, или SO, будет просто SO, как и простейшая формула диоксида дисеры, S2O2. Таким образом, оксид серы и диоксид дисеры, оба являющиеся соединением серы и кислорода, имеют одну и ту же простейшую формулу. Однако их химические формулы, которые выражают количество атомов в каждой молекуле химического соединения, не совпадают.

Mercury — бесплатное программное обеспечение, разработанное Кембриджским центром кристаллографических данных. Первоначально разработано как инструмент визуализации кристаллической структуры. Mercury предоставляет возможность трехмерной визуализации кристаллической структуры, а также рисовании и анализе кристаллической упаковки и межмолекулярных взаимодействий. Текущая версия Mercury может читать файлы типов «.cif», «.mol», «.mol2», «.pdb», «.res», «.sd» и «.xyz». Mercury имеет собственный формат файла с расширением имени файла «.mryx».