TEV-протеаза
Протеаза TEV (от англ. Tobacco Etch Virus — вирус гравировки табака) — высокоспецифичная цистеиновая протеаза вируса гравировки табака. Относится к суперсемейству PA из химотрипсин-подобных протеаз. Благодаря своей высокой специфичности к последовательности часто используется для контролируемого расщепления гибридных белков in vitro и in vivo.[1]
Происхождение
Весь геном вируса гравировки табака кодирует единственный массивный полипротеин (350 кДа), который расщепляется на функциональные блоки тремя протеазами: Р1-протеазой (1 сайт расщепления), HC-Pro (1 сайт расщепления) и TEV-протеазой (7 сайтов расщепления).[2] Нативная протеаза также содержит внутренний участок саморасщепления. Этот участок медленно расщепляется, чтобы инактивировать фермент (физиологическая причина этого неизвестна).
Структура и функции

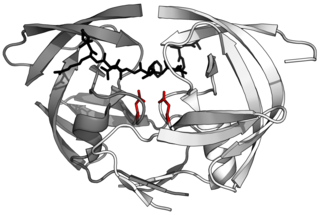
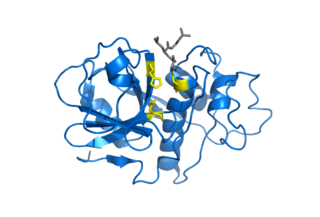
Структура TEV-протеазы была определена с помощью рентгеновской кристаллографии.[3] Фермент состоит из двух β-цилиндров и гибкого C-конца, что показывает структурную гомологию с суперсемейством трипсиновых протеиназ (PA-клан, семейство С4 по классификации MEROPS).[4] Несмотря на гомологию с сериновыми протеазами (такими как трипсин, эластаза, тромбин и т. д.), в протеазе TEV используется цистеин в качестве каталитического центра[5] (как и во многих других вирусных протеазах).
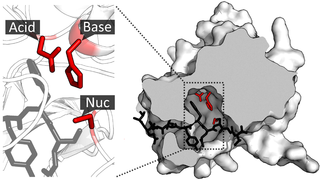
Ковалентный катализ происходит с помощью триады Asp-His-Cys, разделенной между двумя β-цилиндрамим (Asp находится на β1, а His и Cys на β2).[6] Субстрат удерживается в виде β-листа, образуя антипараллельное взаимодействие с бороздкой между двумя цилиндрами и параллельное взаимодействие с C-концом.[7] Таким образом, фермент образует связывающий туннель вокруг субстрата, а взаимодействия боковой цепи обеспечивают специфичность.[3]
Специфичность

Естественная последовательность расщепления была сначала определена путем изучения участков разрезания в нативном полипротеиновом субстрате для повторяющейся последовательности. Консенсусной последовательностью нативного сайта расщепления является ENLYFQ\S (где ‘\’ обозначает расщепляемую пептидную связь).[8] Аминокислотные остатки субстрата нумеруются с Р6 по Р1 до сайта расщепления и Р1’ после вырезанного участка. В ранних работах также проводили оценку расщепления массива схожих субстратов, чтобы определить насколько специфично фермент будет расщеплять нативную последовательность.[9][10]
В исследованиях впоследствии применялось секвенирование расщепляемых субстратов из пула рандомизированных последовательностей для определения предпочтительных паттернов.[11][12] Хотя ENLYFQ\S — это оптимальная последовательность, протеаза активна в большей или меньшей степени на различных субстратах (т. е. проявляет некоторую вариабельность). Наиболее эффективное расщепление происходит в последовательностях наиболее похожих на консенсус EXLYΦQ\φ, где X - любой аминокислотный остаток, Φ - любой большой или гидрофобный остаток среднего размера, а φ - любой небольшой гидрофобный или полярный аминоктслотный остаток.
Специфичность обеспечивается большой площадью контакта между ферментом и субстратом. Протеазы, такие как трипсин, специфичны для одного аминокислотного остатка до и после расщепляемой связи за счет мелкой связывающей щели только с одним или двумя карманами, которые связывают боковые цепи субстрата. И наоборот, вирусные протеазы, такие как TEV-протеаза, имеют длинный C-концевой хвост, который полностью закрывает субстрат для создания связывающего тоннеля. Этот туннель содержит набор связывающих карманов, так что каждая боковая цепь пептидного субстрата (с Р6 до Р1’) связывается с комплиментарным сайтом (с С6 до С1’).[3]
В частности, пептидная боковая цепь P6-Glu контактирует с сетью из трех водородных связей; P5-Asn указывает на растворитель, не создавая специфичных взаимодействий (по этой причине отсутствует консенсус в последовательности субстрата в данном положении); P4-Leu погружает в гидрофобный карман; P3-Tyr удерживает в гидрофобном кармане с короткой водородной связью в конце; P2-Phe также окружен гидрофобнымими остатками, включая поверхность триады гистидина; P1-Gln образует четыре водородные связи; и P1'-Ser только частично заключен в мелкую гидрофобную канавку.[3]
В качестве биохимического инструмента
Одним из основных направлений использования этого фермента является удаление афинных тагов из препаратов очищенного гибридного белка. Причиной использования TEV-протеазы в качестве биохимического инструмента является его высокая специфичность к последовательности. Это делает его относительно нетоксичным in vivo, поскольку узнаваемая им последовательность почти не встречается в белках.[13]
Хотя рациональное конструирование имело ограниченный успех в изменении специфичности протеазы, направленная эволюция использовалась для смены предпочтительного остатка до[14] либо после[15][16] сайта расщепления.
TEV-протеаза имеет ограничения в использовании. Она склонна к дезактивации путем саморасщепления, хотя этого можно избежаь путём мутации S219V во внутреннем сайте разрезания[17]. Протеаза экспрессированная в одиночку плохо растворима; было сделано несколько попыток для улучшения её растворимости путем направленной эволюции и компьютерного моделирования. Кроме того, было показано, что экспрессия может быть улучшена путем слияния с мальтоза-связывающим белком, который повышает растворимость партнера.
Молекулярная масса этого фермента лежит в пределах от 25 до 27 кДа в зависимости от того, какая конструкция используются.
Внешние ссылки
- Полипротеин TEV на UniProt
- TEV-ротеаза на MEROPS
- FAQ по TEV-протеазе на сайте Национального института онкологии (США)
Ссылки
- ↑ Kapust R.B., Waugh D.S. Controlled intracellular processing of fusion proteins by TEV protease (англ.) // Protein Expression and Purification[англ.] : journal. — 2000. — July (vol. 19, no. 2). — P. 312—318. — doi:10.1006/prep.2000.1251. — PMID 10873547.
- ↑ UniProt: TEV polyprotein: P04517. Дата обращения: 11 июля 2017. Архивировано 27 декабря 2016 года.
- ↑ 1 2 3 4 Phan J., Zdanov A., Evdokimov A.G., Tropea J.E., Peters H.K., Kapust R.B., Li M., Wlodawer A., Waugh D.S. Structural basis for the substrate specificity of tobacco etch virus protease (англ.) // Journal of Biological Chemistry : journal. — 2002. — December (vol. 277, no. 52). — P. 50564—50572. — doi:10.1074/jbc.M207224200. — PMID 12377789.
- ↑ Rawlings N.D., Barrett A.J., Bateman A. MEROPS: the database of proteolytic enzymes, their substrates and inhibitors (англ.) // Nucleic Acids Research : journal. — 2012. — January (vol. 40, no. Database issue). — P. D343—50. — doi:10.1093/nar/gkr987. — PMID 22086950. — PMC 3245014.
- ↑ Bazan J.F., Fletterick R.J. Viral cysteine proteases are homologous to the trypsin-like family of serine proteases: structural and functional implications (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 1988. — November (vol. 85, no. 21). — P. 7872—7876. — doi:10.1073/pnas.85.21.7872. — . — PMID 3186696. — PMC 282299.
- ↑ Dougherty W.G., Parks T.D., Cary S.M., Bazan J.F., Fletterick R.J. Characterization of the catalytic residues of the tobacco etch virus 49-kDa proteinase (англ.) // Virology : journal. — 1989. — September (vol. 172, no. 1). — P. 302—310. — doi:10.1016/0042-6822(89)90132-3. — PMID 2475971.
- ↑ Tyndall J.D., Nall T., Fairlie D.P. Proteases universally recognize beta strands in their active sites (англ.) // Chemical Reviews[англ.] : journal. — 2005. — March (vol. 105, no. 3). — P. 973—999. — doi:10.1021/cr040669e. — PMID 15755082.
- ↑ Carrington J.C., Dougherty W.G. A viral cleavage site cassette: identification of amino acid sequences required for tobacco etch virus polyprotein processing (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 1988. — May (vol. 85, no. 10). — P. 3391—3395. — doi:10.1073/pnas.85.10.3391. — . — PMID 3285343. — PMC 280215.
- ↑ Dougherty W.G., Cary S.M., Parks T.D. Molecular genetic analysis of a plant virus polyprotein cleavage site: a model (англ.) // Virology : journal. — 1989. — August (vol. 171, no. 2). — P. 356—364. — doi:10.1016/0042-6822(89)90603-X. — PMID 2669323.
- ↑ Kapust R.B., Tözsér J., Copeland T.D., Waugh D.S. The P1' specificity of tobacco etch virus protease (англ.) // Biochemical and Biophysical Research Communications[англ.] : journal. — 2002. — June (vol. 294, no. 5). — P. 949—955. — doi:10.1016/S0006-291X(02)00574-0. — PMID 12074568.
- ↑ Boulware K.T., Jabaiah A., Daugherty P.S. Evolutionary optimization of peptide substrates for proteases that exhibit rapid hydrolysis kinetics (англ.) // Biotechnology and Bioengineering[англ.] : journal. — 2010. — June (vol. 106, no. 3). — P. 339—346. — doi:10.1002/bit.22693. — PMID 20148412.
- ↑ Kostallas G., Löfdahl PÅ, Samuelson P. Substrate profiling of tobacco etch virus protease using a novel fluorescence-assisted whole-cell assay (англ.) // PLoS ONE : journal. — 2011. — Vol. 6, no. 1. — P. e16136. — doi:10.1371/journal.pone.0016136. — PMID 21267463. — PMC 3022733.
- ↑ Parks T.D., Leuther K.K., Howard E.D., Johnston S.A., Dougherty W.G. Release of proteins and peptides from fusion proteins using a recombinant plant virus proteinase (англ.) // Analytical Biochemistry[англ.] : journal. — 1994. — February (vol. 216, no. 2). — P. 413—417. — doi:10.1006/abio.1994.1060. — PMID 8179197.
- ↑ Engineering of TEV protease variants by yeast ER sequestration screening (YESS) of combinatorial libraries (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 2013. — April (vol. 110, no. 18). — P. 7229—7234. — doi:10.1073/pnas.1215994110. — PMID 23589865.
- ↑ A tobacco etch virus protease with increased substrate tolerance at the P1' position (англ.) // PLoS ONE : journal. — 2013. — Vol. 8, no. 6. — P. e67915. — doi:10.1371/journal.pone.0067915. — PMID 23826349.
- ↑ Intracellular detection and evolution of site-specific proteases using a genetic selection system (англ.) // Appl. Biochem. Biotechnol. : journal. — 2012. — March (vol. 166, no. 5). — P. 1340—1354. — doi:10.1007/s12010-011-9522-6. — PMID 22270548.
- ↑ Kapust R.B., Tözsér J., Fox J.D., Anderson D.E., Cherry S., Copeland T.D., Waugh D.S. Tobacco etch virus protease: mechanism of autolysis and rational design of stable mutants with wild-type catalytic proficiency (англ.) // Protein Eng. : journal. — 2001. — December (vol. 14, no. 12). — P. 993—1000. — doi:10.1093/protein/14.12.993. — PMID 11809930.